Voraussetzungen
Beginnen Sie mit diesem Experiment erst dann, wenn Sie die grundlegenden Experimente (pipettieren) und die einfachen molekularbiologischen Experimente (u.a Agarosegelelektrophorese, Restriktionsverdau) erfolgreich durchgeführt haben. Die PCR liefert Ihnen bei unsachgemäßer Handhabung negative Resultate.
Sie benötigen
- einen PCR-Cycler mit heizbarem Deckel (z.B. Personal Cycler von Eppendorf)
- chromosomale DNA aus Bakterien
- PCR Reagenzien
- Enzympipette zum genauen Pipettieren von 0,5µl-Volumina
- Ausstattung für die Agarosegelelektrophorese
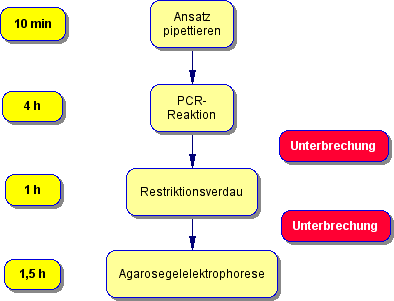
Zeitbedarf: Sie benötigen etwa 10-20 min für das Pipettieren des PCR Ansatzes. Für die PCR benötigt Sie etwa 4 Stunden. Für die Agarosegelelektrophorese sind weitere 1-1,5 Stunden einzuplanen.
Folgeexperimente: Klonierung, Transformation, Nukleotidsequenzierung
Aufgaben
- Isolieren Sie chromosomale DNA aus verschiedenen Mikroorganismen (E. coli, Bacillus subtilis).
- Führen Sie mit dieser DNA als Vorlage eine pCR auf deren 16S rRNA Gene durch.
- Analysieren Sie die PCR-Amplifikate in der Agarosegelelektrophorese.
Lernziele
Sie lernen an einem einfachen Beispiel die mit einem Nobel-Preis ausgezeichnete PCR-Technik und das RFLP-Fingerprinting kennen. Dabei werden einige Prinzipien beschrieben, die Ihnen die Anwendung dieser Methoden auf andere Fragestellungen erleichtern werden.
Probleme
| Problem | Lösungsansatz |
| Es ist kein PCR-Produkt erkennbar. | Haben Sie alle Komponenten im Ansatz vorgelegt? Stimmt das Temperaturprogramm? Stimmt die Primer-Hybridisierungstemperatur (Annealing Temperatur)? War die Taq-Polymerase aktiv? Geben Sie eine Kontrolle in Ihren Ansatz, um den PCR-Ansatz auf hemmende Komponenten zu analysieren. |
| In der Front ist eine sehr helle Bande, sonst aber kein PCR-Produkt erkennbar. | Sie haben vermutlich die Primer überkonzentriert. Verdünnen Sie mindestens 1:10 oder 1:50 mit sterilem Wasser. |
| Es sind mehr als eine Bande erkennbar. | Vermutlich kam es zu unspezifischen Reaktionen. Erhöhen Sie die Annealing Temperatur und/oder variieren Sie die Mg2+ Konzentrationen. |
Sicherheit
Der Versuch hat keine erkennbaren Sicherheitsrisiken für den Experimentator oder die Umwelt, so lange Sie mit der chromosomalen DNA aus definierten, nicht pathogenen Bakterien arbeiten und die angegebenen Primer verwenden. Beachten Sie auch die Sicherheitshinweise zur Agarosegelelektrophorese, wenn Sie die PCR-Produkte nachweisen.
| Tabelle 1: Sicherheitsdatenblätter als pdf-Dateien | |
| Abkürzungen, Gefahrensymbole, R+S-Sätze | 100 KB |
Tipps und Hinweise
- Achten Sie auf exakte Volumina und richtige Konzentrationen der PCR-Ansatzkomponenten. Kalibrieren Sie eventuell Ihre Mikroliterpipetten!
- Mischen Sie die Testkomponenten. Zentrifugieren Sie das PCR-Reaktionsgefäß notfalls kurz in einer Tischzentrifuge an, damit sich die gesamte Flüssigkeit am Boden des Gefäßes sammelt.
- Setzen Sie die PCR-Reaktionsgefäße in die Mitte des Heizblocks; vermeiden Sie nach Möglichkeit die Randbereiche.
- Nutzen Sie ein PCR-Gerät mit heizbarem Deckel und keine Wasserbäder für die Temperaturzyklen.
- Verwenden Sie nur qualitativ hochwertige DNA-Polymerasen. Es gibt große qualitative Unterschiede.
- Für sehr kritische Reaktionen sollten Sie die langsameren und teueren "proof-reading" Polymerasen verwenden.
- Je länger das zu erwartende PCR-Produkt ist, desto länger sollte die Polymerisationszeit sein.
Unterlagen
Die Zahlen geben die Dateigrößen in KB wieder. Zum Lesen der pdf-Dateien benötigen Sie den Adobe Acrobat Reader (einige Dateien wurden für Version 7 oder besser optimiert!). Zum Betrachten der ppt-Dateien benötigen Sie Microsofts PowerPoint. Deren Ausdruck als pdf-Datei erlaubt Ihnen die Herstellung von Folien für den Tageslichtprojektor.
Tipp: Laminieren Sie solche Seiten, die Sie häufiger als Arbeitsunterlagen nutzen möchten.
Fragen
- Warum empfiehlt sich ein heizbarer Deckel für die Kammer der PCR-Gefäße?
- Warum sind Magnesiumionen im PCR-Ansatz von Bedeutung?
- In welchem Zusammenhang steht die Hybridisierungstemperatur eines Primers zu seinem GC-Gehalt?
- Warum können Sie doppelsträngige DNA zu Einzelsträngen aufschmelzen?
- Warum können PCR-Primer nicht an doppelsträngige DNA binden?
- Was ist eine "proofreading" Aktivität?
